RNAs in der Zelle sind in hohem Maße mit Proteinen dekoriert, die ihre Funktion steuern. Wir sind besonders daran interessiert zu verstehen, wie die Struktur einer RNA die Erkennung durch Proteine beeinflusst und sich dadurch letztlich auf zelluläre Entscheidungen auswirkt. Um neue funktionelle RNA-Strukturen zu identifizieren, nutzen wir das Konzept der evolutionären Konservierung.
Die evolutionäre Konservierung von Sequenzen innerhalb einer mRNA weist auf eine funktionelle Bedeutung hin. Von besonderer Bedeutung ist eine hohe Konservierung der Protein-kodierenden Sequenzen. Die Sequenzkonservierung innerhalb untranslatierter Regionen einer mRNA ist in der Regel gering; wenn sie auftritt, impliziert sie eine Funktion der entsprechenden Sequenz in der post-transkriptionellen Regulation. mRNAs sind, wie andere RNA-Moleküle auch, in der Lage, komplexe dreidimensionale Strukturen zu bilden. Die evolutionäre Konservierung einer mRNA-Faltung ist daher ebenfalls ein Hinweis auf eine entsprechende Funktion.
Mithilfe einer bioinformatischen Vorhersage der mRNA-Faltung und evolutionärer Konservierung haben wir neue Bindungsstellen für Roquin-Proteine identifiziert. Roquin-Proteine erkennen mRNAs struktur-spezifisch und sequenzunabhängig. Die Bindung von Roquin an solche so genannten CDEs (constitutive decay elements) beschleunigt den mRNA-Abbau (Abb. 1).
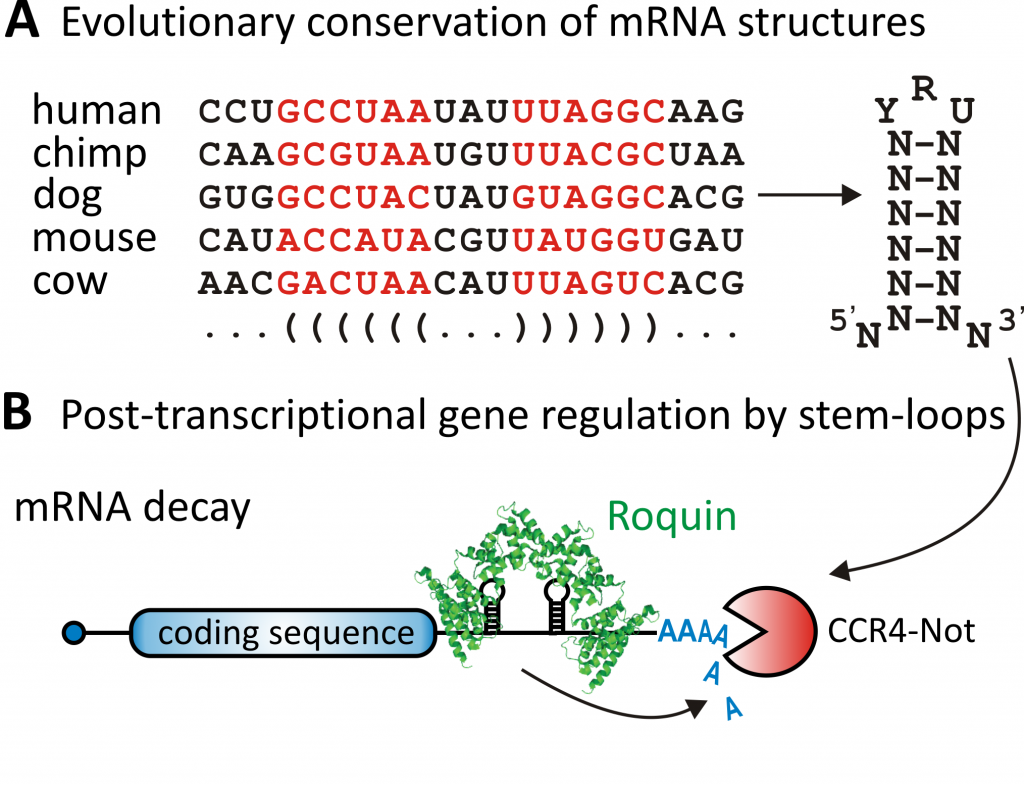
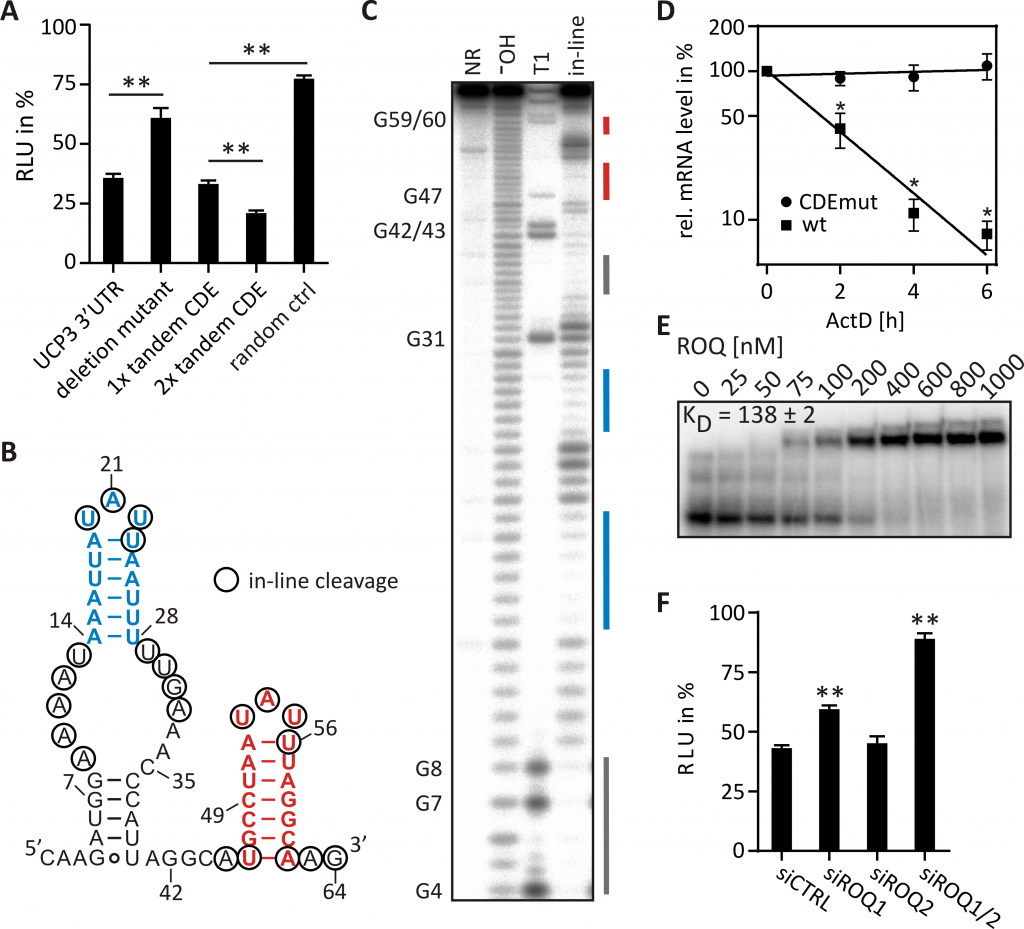
So entdeckten wir beispielsweise ein Tandem-CDE in der 3′-UTR der UCP3 mRNA (Abb. 2). Struktur- und Mutationsanalysen bestätigten die Bedeutung der beiden Stammschleifen für die mRNA-Destabilisierung. Die Roquinbindung wurde in vitro, die post-transkriptionelle Regulation in vivo bestätigt. Die eingehende Mutationsanalyse des Tandem-CDEs ermöglichte es uns, die Bindungspräferenzen von Roquin besser zu verstehen und eine Vielzahl anderer CDEs in bisher unbekannten Zielgenen zu entdecken. Außerdem untersuchten wir die Interaktion von Roquin mit anderen RNA-bindenden Proteinen, die um die mRNA-Bindung konkurrieren. Wir entdeckten, dass AU-reiche CDEs von Roquin als Stammschleife und von dem Konkurrenten AUF1 in ihrer linearen Form erkannt werden, was auf eine Doppelfunktion im mRNA-Abbau in Abhängigkeit von ihrem Faltungsstatus hindeutet.
Mit unseren derzeitigen Forschungsarbeiten wollen wir uns ein vollständiges Bild von den Roquin-Bindungspräferenzen und der Hierarchie der adressierten Zielgene machen. Außerdem analysieren wir die Funktion anderer RNA-Strukturen bei der posttranskriptionellen Genregulation mit Hilfe von massiv parallelen Reporter-Assays und SHAPE-Map.
