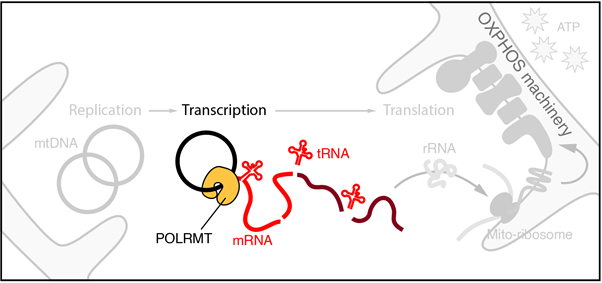
Die Gruppe untersucht schwerpunktmäßig, wie die Genexpression in Säugetiermitochondrien reguliert wird. Mitochondrien sind Organellen, die dynamische Netzwerke in eukaryotischen Zellen bilden und vor allem für ihre Rolle im Energiestoffwechsel bekannt sind, da sie den Großteil des zellulären Adenosintriphosphats (ATP) durch oxidative Phosphorylierung synthetisieren. Mitochondrien sind auch wichtige Signalvermittler, die den programmierten Zelltod (Apoptose), die Stressantwort und angeborene Immunreaktionen koordinieren.
Neben ihrer Rolle im Zellstoffwechsel und in der Signalübertragung enthält das Mitochondriennetzwerk in einer Säugetierzelle typischerweise mehrere tausend Kopien eines zirkulären, doppelsträngigen DNA-Genoms (mtDNA) – das genetische Erbe einer uralten endosymbiotischen Beziehung. Dieses “zweite Genom” kodiert einzigartige Proteinkomponenten der oxidativen Phosphorylierungsmaschinerie und ist für die mitochondriale Funktion und das Überleben der Zelle unerlässlich. Defekte in der Art und Weise, wie die mtDNA kopiert und exprimiert wird, sind in hohem Maße am Alterungsprozess sowie an mehreren verheerenden und häufig vererbten menschlichen Krankheiten beteiligt, die allgemein als mitochondriale Krankheiten bezeichnet werden. Derzeit gibt es keine bekannte Behandlung für mitochondriale Erkrankungen, und ein Faktor, der zu dieser schlechten Prognose beiträgt, ist ein unzureichendes grundlegendes Verständnis darüber, wie die mitochondriale Genexpression in lebenden Zellen sowohl auf basaler Ebene als auch zwischen Zelltypen mit unterschiedlichen energetischen Anforderungen reguliert wird.
Unsere Arbeit verwendet in erster Linie biochemische Ansätze mit gereinigten rekombinanten Proteinen und Nukleinsäuresubstraten, um molekulare Schlüsselereignisse in vitro zu rekonstruieren, und kombiniert dies mit genetischen Ansätzen in kultivierten Zelllinien, um den in vivo-Kontext unserer Arbeit herzustellen. Langfristiges Ziel der Gruppe ist es zu verstehen, wie die mitochondriale Genexpression auf der Ebene der naszierenden Transkription reguliert wird und wie der mitochondriale RNA-Stoffwechsel zu sub-mitochondrialen Strukturen, den so genannten RNA-Granula, führt. Dadurch hoffen wir, die Pathogenese mitochondrialer Erkrankungen auf genetischer Ebene besser zu verstehen und Angriffspunkte für Therapien zu finden.
Ausgewählte Publikationen:
Tan, B.G., Gustafsson, C.M. & Falkenberg, M. Mechanisms and regulation of human mitochondrial transcription. Nat Rev Mol Cell Biol. (2023). https://doi.org/10.1038/s41580-023-00661-4
Tan, B.G., Mutti, C.D.*, Shi, Y.*, Xie, X., Zhu, X., Silva-Pinheiro, P., Menger, K.E., Díaz-Maldonado, H., Wei, W., Nicholls, T.J., Chinnery P.F., Minczuk, M., Falkenberg, M., Gustafsson, C.M. (2022). The human mitochondrial genome contains a second light strand promoter. Mol Cell 82, 3646-3660.e9. https://doi.org/10.1016/j.molcel.2022.08.011. Open access. (*equal contributions)
Zhu, X.*, Xie, X.*, Das, H.*, Tan, B.G., Shi, Y., Al-Behadili, A., Peter, B., Motori, E., Valenzuela, S., Posse, V., Gustafsson, C.M., Hällberg, B.M., Falkenberg, M. (2022). Non-coding 7S RNA inhibits transcription via mitochondrial RNA polymerase dimerization. Cell 185, 2309-2323.e24. https://doi.org/10.1016/j.cell.2022.05.006. Open Access. (*equal contributions)
Tan, B.G., Wellesley, F.C., Savery, N.J., and Szczelkun, M.D. (2016). Length heterogeneity at conserved sequence block 2 in human mitochondrial DNA acts as a rheostat for RNA polymerase POLRMT activity. Nucleic Acids Res 44, 7817–7829. https://doi.org/10.1093/nar/gkw648. Open access.
