RNA-Viren müssen Kontrollsignale für die Replikation des Genoms, die Transkription subgenomischer mRNAs und die virale Proteinsynthese in den begrenzten Raum ihrer Genome integrieren. Diese Prozesse werden häufig durch RNA-Strukturen gesteuert, die von Virus- und Wirtsproteinen erkannt werden und eine effiziente Vermehrung des Virus in der Zelle ermöglichen.
Das RNA-Genom von SARS-CoV-2 trägt essentielle RNA-Strukturen an seinen 5′- und 3′-Enden sowie in der Frameshifting-Region (Abb. 1). Obwohl diese Strukturen unter den Betacoronaviren hoch konserviert sind, ist die Funktion der meisten von ihnen unbekannt.
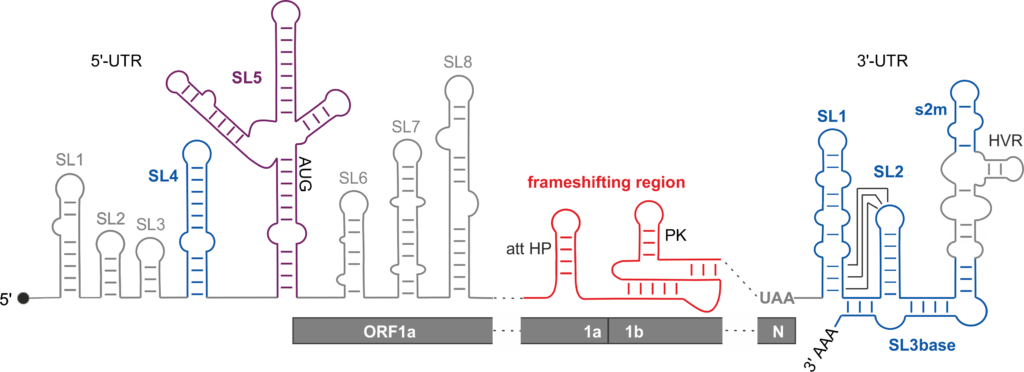
In Kollaboration haben wir die RNA-Faltung dieser funktionellen SARS-CoV-2-RNA-Strukturen analysiert und entwickeln niedermolekulare Liganden zur RNA-Hemmung als antivirale Mittel. In der aktuellen Forschung untersuchen wir die Struktur-Funktions-Beziehung einzelner RNA-Faltungen und die Art der Interaktion mit kleinen Molekülen und Proteinen.
